教員個人(研究室)ページ
ゲノム微生物学研究室
Laboratory of Genome Microbiology
担当教員: 島田 友裕 教授/博士(工学)

<研究略歴>
2008年法政大学大学院工学研究科物質化学専攻博士課程修了。
法政大学マイクロ・ナノテクノロジー研究所 博士研究員、
東京工業大学 資源化学研究所 助教を経て、2017年より現職
<主な担当科目>
生化学II、応用生化学、生化学・物理化学実験
<研究室所在>
第一校舎3号館3階308号室
研究テーマ
「遺伝子発現の仕組みの解明から、微生物の理解・応用を目指しています」
現在までに数万種類を越える微生物のゲノム配列が決定され、1つの生物を構築する遺伝子の全体像が明らかとなりました。しかし、ゲノム上にはなお、生理機能未知の遺伝子が多く、加えて、発現され利用される遺伝子の選択システムについては不明な点が多いです。全遺伝子の機能情報とゲノムの選択的発現の包括制御機構の解明は、21世紀生命科学の重要課題です。
私たちは、1つの生物としては個々の遺伝子機能の知見が最も蓄積している大腸菌をモデル生物として、ゲノムから発現させる遺伝子の選択や強度を決定するための分子機構について研究しています。大腸菌ゲノムにおける全転写制御因子(シグマ因子が全7種類、転写因子が約300種類)の機能および役割を解析しており、終局的には1つの生物丸ごとのゲノム転写包括制御機構の理解を目指しています。
特に、転写制御因子のゲノム上結合領域を網羅的に同定するために、Genomic SELEX法 (gSELEX法)という解析手法を開発し、本手法を持ちいた独自の研究戦略を強みとしています。試験管の中で転写制御因子と断片化されたゲノムDNAを混合し、形成した転写制御因子-DNAの複合体を精製し、そのDNAの塩基配列を分析することで、転写制御因子の直接的なゲノム上の結合領域を同定する手法です。
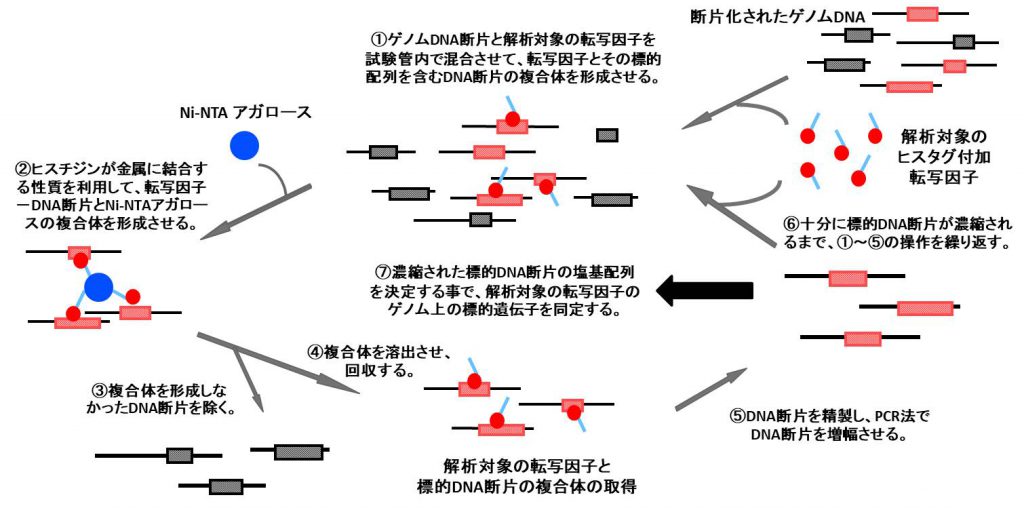
転写制御因子の制御ネットワークやその役割を理解することで、ゲノムから遺伝子が利用される仕組みを分子レベルで明らかにすると共に、共同研究等を通じてそれらの知見の応用展開を試みています。
・研究室メンバー:大学院前期2年1名:大学院前期1年5名:学部4年6名;学部3年10名;博士研究員1名
キーワード:ゲノム、大腸菌、転写制御、転写因子、シグマ因子、RNAポリメラーゼ、Genomic SELEX (gSELEX)、転写制御ネットワーク、遺伝子発現、生分解性プラスチック、自然環境適応機構
研究業績
Shimada, T. Furuhata, S. and Ishihama, A. The whole set of constitutive promoters for RpoN sigma factor and the regulatory role of its enhancer protein NtrC in Escherichia coli K-12. Microbial Genomics. in press (2021)
Ishihama, A. and Shimada, T. Hierarchy of transcription factor network in Escherichia coli K-12: H-NS mediated genome silencing and anti-silencing by global regulators. FEMS Microbiology Reviews. in press (2021)
Shimada, T., Ogasawara, H., Kobayashi, I., Kobayashi, N. and Ishihama, A. Single-target regulators constitute the minority group of transcription factors in Escherichia coli K-12. Frontiers in Microbiology. 12, 697803 (2021)
Shimada, T., Nakazawa, K., Tachikawa, T., Saito, N., Niwa, T., Taguchi, H. and Tanaka, K. Acetate overflow metabolism regulates a major metabolic shift after glucose depletion in Escherichia coli. FEBS Lett. 595, 2047-2056 (2021)
Sharma, M., Abayakoon, P., Epa, R., Jin, Y., Lingford, JP., Shimada, T., Nakano, M., Mui, JWY., Ishihama, A., Boddard-Borger, ED., Davies GJ. and Williams, SJ. Molecular basis of sulfosugar selectivity in sulfoglycolysis. ACS Cent Sci. 7, 476-487 (2021)
Anzai, T., Imamura, S., Ishihama, A. and Shimada, T. Expanded roles of pyruvate- sensing PdhR in transcription regulation of the Escherichia coli K-12 genome: fatty acid catabolism and cell motility. Microbial Genomics. 6, mgen000442 (2020)
Ogasawara, H., Ishizuka, T., Hotta, S., Aoki, M., Shimada, T. and Ishihama, A. Novel regulators of the csgD gene encoding the master regulator of biofilm formation in Escherichia coli K-12. Microbiology. 166, 880-890 (2020)
Shimizu, T., Yasuda, R., Mukai, Y., Tanoue, R., Shimada, T., Imamura, S., Tanaka, K., Watanabe, S. and Masuda, T. Proteomic analysis of heme-binding protein from Arabidopsis thaliana and Cyanidioschyzon merolae. Philosophical transactions of the royal society B. 375, 20190488 (2020)
Ogasawara, H., Ishizuka, T., Yamaji, K., Kato, Y., Shimada, T. and Ishihama, A. Regulatory role of pyruvate-sensing BtsSR in biofilm formation by Escherichia coli K-12. FEMS Microbiology letters. 366, pii: fnz251. doi: 10.1093/femsle/fnz251. (2019)
Shimada, T., Yokoyama, Y., Anzai, T., Yamamoto, K. and Ishihama, A. Regulatory role of PlaR (YiaJ) for plant utilization in Escherichia coli K-12. Scientific Reports. 9, 20415. doi: 10.1038/s41598-019-56886-x. (2019)
Yoshida, H., Wada, A., Shimada, T., Maki, Y. and Ishihama, A. Coordinated hibernation of transcription apparatus and translation machinery upon transfer of Escherichia coli growth into stationary phase. Frontiers in Genetics. doi: 10.3389/fgene.2019.01153. (2019)
Shimizu, T., Kacprzak, S., Mochizuki, N., Nagatani, A., Watanabe, S., Shimada, T., Tanaka, K., Hayashi, Y., Arai, M., Leister, D., Okamoto, H., Terry, M. and Masuda, T. The regrograde signaling protein GUN1 regulates tetrapyrrole biosynthesis. Proceedings of American National Academy of Science, U.S.A. 116, 24900-24906 (2019)
Akanuma, G., Tagana, T., Sawada, M., Suzuki, S., Shimada, T., Tanaka, K., Kawamura, F. and Kato-Yamada, Y. C-terminal regulatory domain of the ε subunit of Fo F1 ATP synthase enhances the ATP-dependent H+ pumping that is involved in the maintenance of cellular membrane potential in Bacillus subtilis. Microbiologyopen. 8, e00815 (2019)
Shimada, T., Yamamoto, K., Nakano, M., Watanabe, H., Schleheck, D. and Ishihama, A. Regulatory role of CsqR (YihW) in transcription of the genes for catabolism of the anionic sugar sulfoquinovose (SQ) in Escherichia coli K-12. Microbiology. 165, 78-89 (2019)
Shimada, T., Ogasawara, H. and Ishihama, A. Genomic SELEX screening of regulatory targets of Escherichia coli transcription factors. Methods Mol Biol. 1837, 49-69 (2018)
Yoshida, H., Shimada, T. and Ishihama, A. Coordinated hibernation of transcriptional and translational apparatus during growth transition of Escherichia coli to stationary phase. mSystems. 3, e00057-18 (2018)
Shimada, T., Ogasawara, H. and Ishihama, A. Single-target regulators form a minor group of transcription factors in Escherichia coli. Nucleic Acids Res. 46, 3921-3936 (2018)
Yamamoto, K., Yamanaka, Y., Shimada, T., Sarkar, P., Yoshida, M., Bharadwaj, N., Watanabe, H., Taira, Y., Chatterji, D. and Ishihama A. Altered distribution of RNA polymerase lacking the omega subunit within the prophages along the Escherichia coli K-12 genome. mSystems. 3, e00172-17. (2018)
Shimada, T., Momiyama, E., Yamanaka, Y., Watanabe, H., Yamamoto, K. and Ishihama, A. Regulatory role of XynR (YagI) in catabolism of xylonate in Escherichia coli K-12. FEMS Microbiology Letters. 364, doi: 10.1093/femsle/fnx220. (2017)
Shimada, T., Tanaka, K. and Ishihama, A. The whole set of the constitutive promoters recognized by four minor sigma subunits of Escherichia coli RNA polymerase. PLoS ONE. 12, e0179181 (2017)
Sugino, H., Usui, T., Shimada, T., Nakano, M., Ogasawara, H., Ishihama, A. and Hirata, A. A structural sketch of RcdA, a transcription factor controlling the master regulator of biofilm formation. FEBS Letters. doi:10.1002/1873-3468.12713 (2017)
Sugimoto, R., Saito, N., Shimada, T. and Tanaka, K. Identification of YbhA as the pyridoxal 5′-phosphate (PLP) phosphatase in Escherichia coli: Importance of PLP homeostasis on the bacterial growth. J. Gen. Appl. Microbiol.63, 362-368 (2017)
Kobayashi, I., Watanabe, S., Kanesaki, Y., Shimada, T., Yoshikawa, H. and Tanaka, K. Conserved two-component Hik34-Rre1 module directly activates heat-stress inducible transcription of major chaperone and other genes in Synechococcus elongatus PCC 7942. Mol Microbiol. 104, 260-277 (2017)